工程化MHC II类四聚体通过增强CD4结合来提高抗原特异性T细胞的检测灵敏度
T细胞通过识别与主要组织相容性复合物(MHC)分子结合的特异性肽抗原,在细胞介导免疫应答中发挥核心调控作用。荧光标记的肽:MHC I类(P:MHCI)四聚体已成为流式细胞术中鉴定抗原特异性CD8⁺ T细胞的标准工具,但其向CD4⁺ T细胞检测领域的拓展仍面临技术瓶颈。
- 最新进展
- 产品信息
MHC class II tetramers engineered for enhanced binding to CD4 improve detection of antigen-specific T cells
研究背景
T细胞通过识别与主要组织相容性复合物(MHC)分子结合的特异性肽抗原,在细胞介导免疫应答中发挥核心调控作用。荧光标记的肽:MHC I类(P:MHCI)四聚体已成为流式细胞术中鉴定抗原特异性CD8⁺ T细胞的标准工具,但其向CD4⁺ T细胞检测领域的拓展仍面临技术瓶颈。这一局限性可能源于CD4分子与MHC II类分子间较低的结合亲和力。本研究通过定向进化技术,成功构建了具有增强CD4结合能力的工程化P:MHC II类四聚体,显著提升了抗原特异性CD4⁺ T细胞的检测效能。实验结果显示,在多种肽抗原免疫的小鼠模型中,新型四聚体检测到的抗原特异性CD4⁺ T细胞频率较传统试剂提升近两倍,揭示了其突破传统检测阈值的能力。该技术突破为解析T细胞免疫应答的精细图谱提供了革新性工具,有望深化对T细胞库动态特征及免疫调控机制的理解。
论文标题
Engineered MHC class II tetramers with enhanced CD4 binding affinity enable superior detection of antigen-specific T cells

研究结果
结果1:定向进化筛选获得与CD4结合增强的I-Ab突变分子
研究团队假设,CD4分子与MHC II类分子间固有的低亲和力可能导致传统P:MHC II类四聚体无法有效检测携带低亲和力T细胞受体(TCR)的T细胞。为突破这一技术瓶颈,研究采用定向进化策略对C57BL/6(B6)小鼠来源的I-Ab MHC II类分子进行改造,旨在获得具有增强CD4结合能力的突变体。
基于上述假设,研究团队构建了编码P5R10抗原肽-I-Abβ链融合蛋白(含E137、V142、I148和L158位点随机突变)的质粒文库,并通过Flippase识别靶位点(FRT)介导的重组技术,将突变文库导入包含I-Abα链及单个FRT位点的CHO细胞系。该系统确保每个CHO细胞表达单一突变型I-Abβ链,形成具有独特氨基酸组合的P5R:I-Ab异源二聚体。随后,利用链霉亲和素-荧光团标记的小鼠CD4四聚体对突变文库进行多轮流式分选,通过磁珠富集CD4结合阳性细胞。经三轮筛选后,成功分离出稳定表达CD4高亲和力突变体的CHO克隆。基因测序证实,富集克隆携带V142I、I148Y及L158D三重突变(命名为I-Ab-4E分子),而野生型P5R:I-Ab分子表达细胞未见结合信号。
进一步功能验证显示,相较于传统P5R:I-Ab四聚体,P5R:I-Ab-4E四聚体对表达低亲和力B3K508 TCR(KD=93 µM)的转基因CD4⁺ T细胞结合能力显著提升,其阳性染色信号接近B6小鼠多克隆CD4⁺ T细胞背景水平。值得注意的是,P5R:I-Ab-4E四聚体并未非特异性结合B6小鼠T细胞库中的绝大多数多克隆T细胞,表明其结合特异性未因亲和力增强而改变。
为验证CD4依赖性,研究采用CRISPR/Cas9技术对B3K508 T细胞进行CD4基因敲除。结果显示,CD4⁻亚群完全丧失与P5R:I-Ab-4E四聚体的结合能力,而残留CD4⁺细胞仍保持结合活性。这一结果明确证实,P5R:I-Ab-4E四聚体与低亲和力TCR的结合增强严格依赖于CD4分子的参与,排除了非特异性相互作用的可能性。
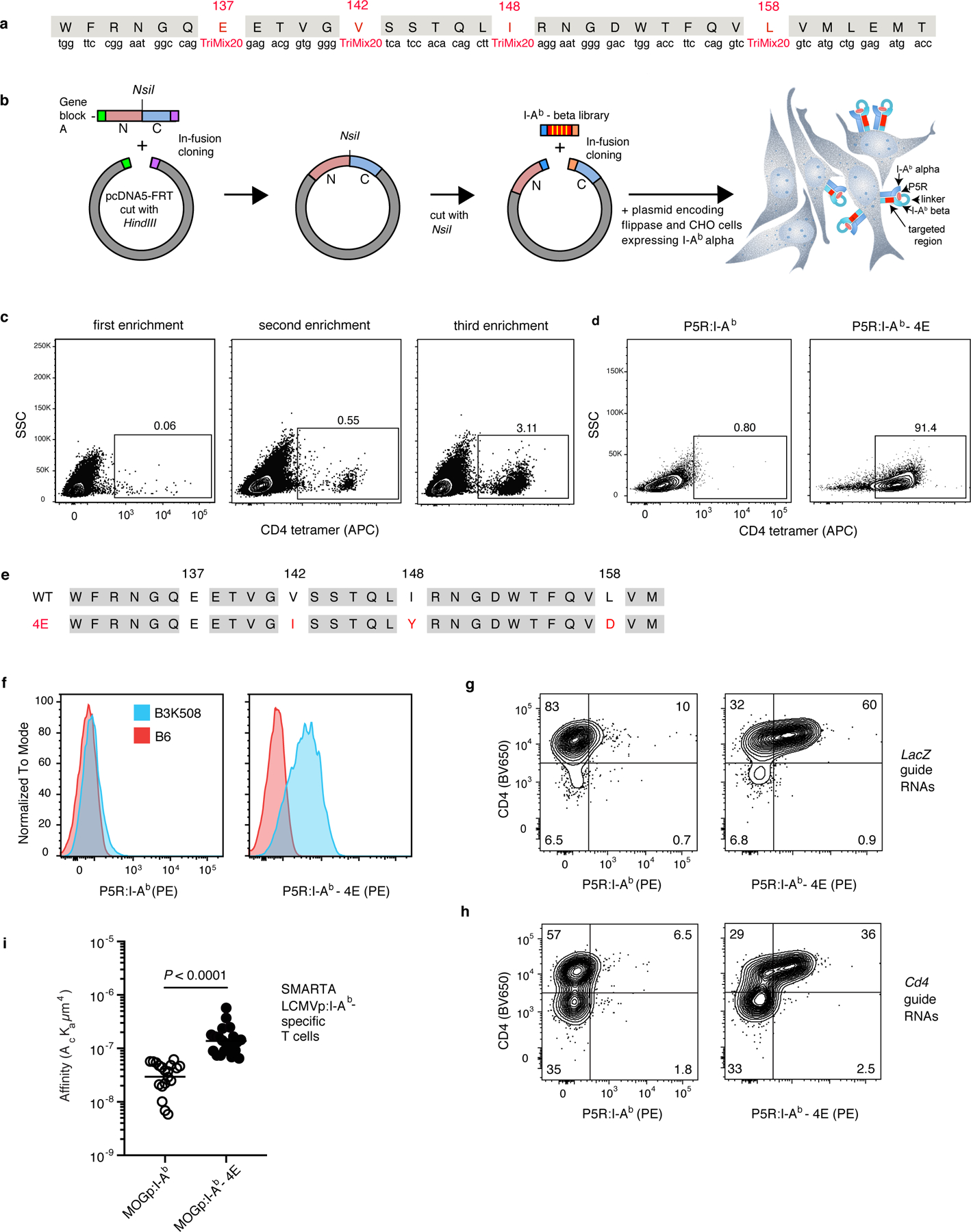
Fig1. 鉴定与CD4分子结合力更强的的I-Ab分子
结果2:工程化四聚体显著提升多品系小鼠抗原特异性CD4+ T细胞检测效能
在金黄色葡萄球菌肽段(P5R、2W、OVA、GP66)及自身抗原(MOGp)免疫的B6小鼠模型中,传统I-Ab四聚体检测到的脾脏与淋巴结来源抗原特异性CD4⁺效应T细胞数量分别为5600、85000、33000、12000或1200个细胞。这种数量差异可能与不同表位特异性初始T细胞的数量差异有关,而MOGp:I-Ab四聚体结合群体的低反应性则反映了中枢免疫耐受机制对自身反应性T细胞的清除作用。相较之下,采用I-Ab-4E工程化四聚体的检测体系使各免疫组效应T细胞检出数量显著提升:P5R、2W、OVA、GP66及MOGp免疫组分别达到9700、110000、4600、63000或5200个细胞,较传统四聚体检测效率提升1.3-4.2倍(平均提升1.7倍)。流式表型分析证实,超过90%的I-Ab或I-Ab-4E四聚体结合细胞呈现CD44^hi效应记忆表型,表明工程化四聚体优先识别活化状态的抗原特异性T细胞。
为验证该技术的跨品系适用性,研究将V142I、I148Y及L158D三重突变(4E突变)分别引入BALB/c小鼠来源的I-Ad分子及NOD小鼠来源的I-Ag7分子,构建OVA3:I-Ad-4E及P31:I-Ag7-4E工程化四聚体。在BALB/c小鼠OVA肽/CFA免疫模型中,OVA3:I-Ad-4E四聚体检出效应T细胞数量较野生型提升2.1倍(12000 vs. 6000个细胞);在NOD小鼠P31肽/CFA免疫模型中,P31:I-Ag7-4E四聚体检出效率提升1.4倍(1400000 vs. 1000000个细胞)。值得注意的是,P31表位因具有高丰度初始T细胞库而呈现强免疫应答,进一步凸显了工程化四聚体在低频抗原特异性T细胞检测中的优势。上述结果表明,4E突变策略可有效提升不同遗传背景小鼠MHC II类分子四聚体的检测灵敏度,为跨品系免疫学研究提供了通用型技术解决方案。
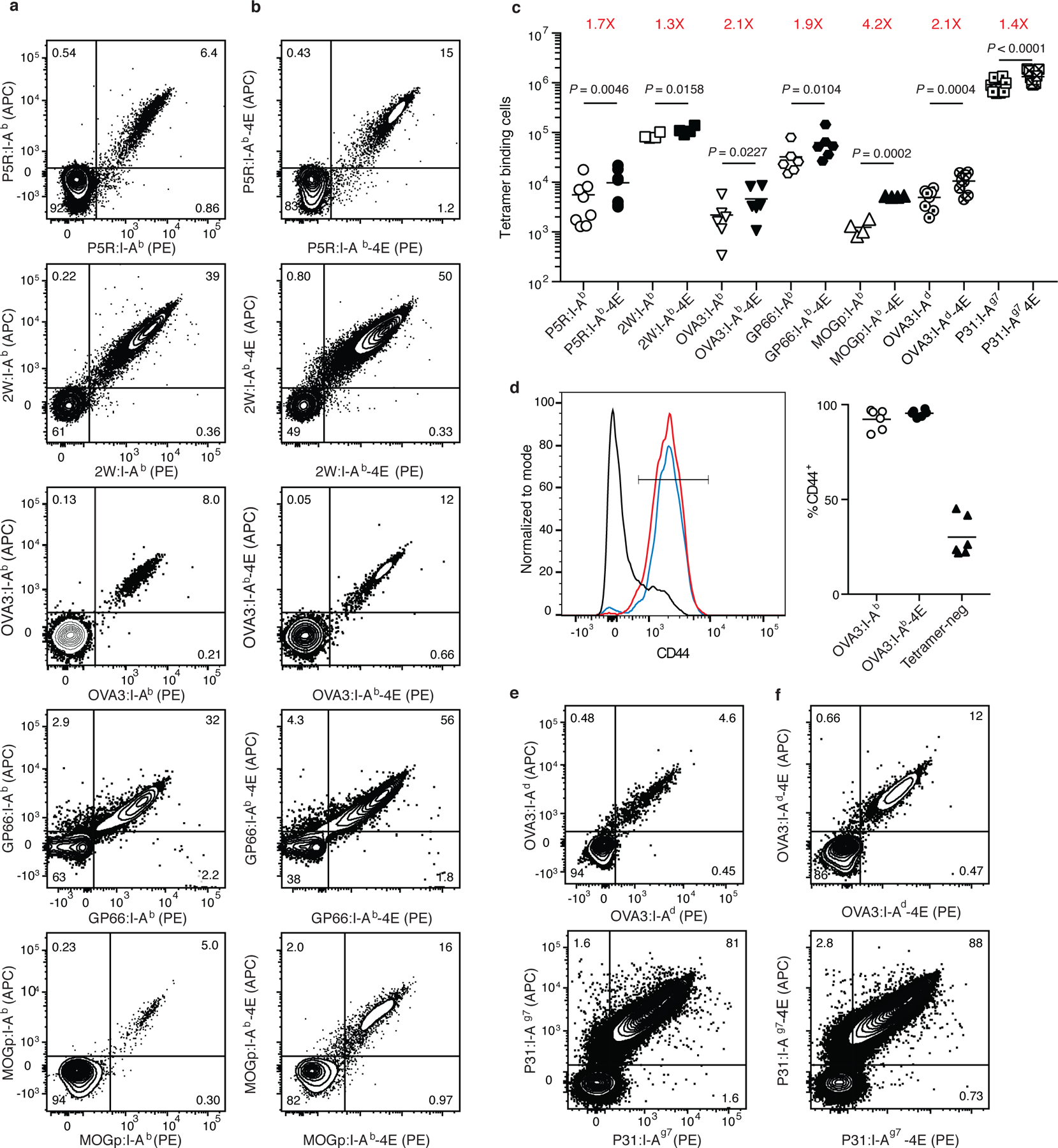
Fig2. 用p:I-Ab -4E四聚体检测免疫小鼠的多克隆CD4+ T细胞
结果3:工程化四聚体严格保持抗原肽特异性识别特性
为验证p:I-Ab-4E四聚体的肽段识别特异性,研究设计了交叉验证实验。基于TCR信号通路驱动抗原特异性幼稚T细胞增殖并上调CD44表达的生物学特性,研究分别采用P5R及MOGp肽段免疫B6小鼠,预期在P5R免疫组中观察到扩增的CD44^hi P5R:I-Ab-4E四聚体结合细胞与低水平CD44^lo MOGp:I-Ab-4E结合细胞共存,而MOGp免疫组呈现相反模式。实验结果完全符合预期:P5R免疫组呈现显著的CD44^hi P5R:I-Ab-4E四聚体结合细胞扩增(占四聚体结合细胞的89±3%),而MOGp:I-Ab-4E结合细胞则维持CD44^lo幼稚表型(占11±2%);MOGp免疫组则检测到大量CD44^hi MOGp:I-Ab-4E结合细胞(87±4%)及少量P5R:I-Ab-4E结合细胞(13±3%)。
关键性交叉染色实验显示,在P5R免疫小鼠中,P5R:I-Ab-4E四聚体结合细胞与MOGp:I-Ab-4E四聚体无交叉反应;MOGp免疫小鼠中亦未检测到跨表位四聚体结合现象。进一步功能验证表明,仅在注射相应抗原肽段的小鼠体内观察到T细胞活化信号(如CD69上调及细胞因子分泌),而无关肽段四聚体结合细胞始终处于静息状态。这些交叉验证实验证实,p:I-Ab-4E四聚体严格识别抗原肽特异性T细胞克隆,其结合反应具有严格的肽段依赖性,排除了非特异性结合或MHC分子多态性导致的背景信号干扰。
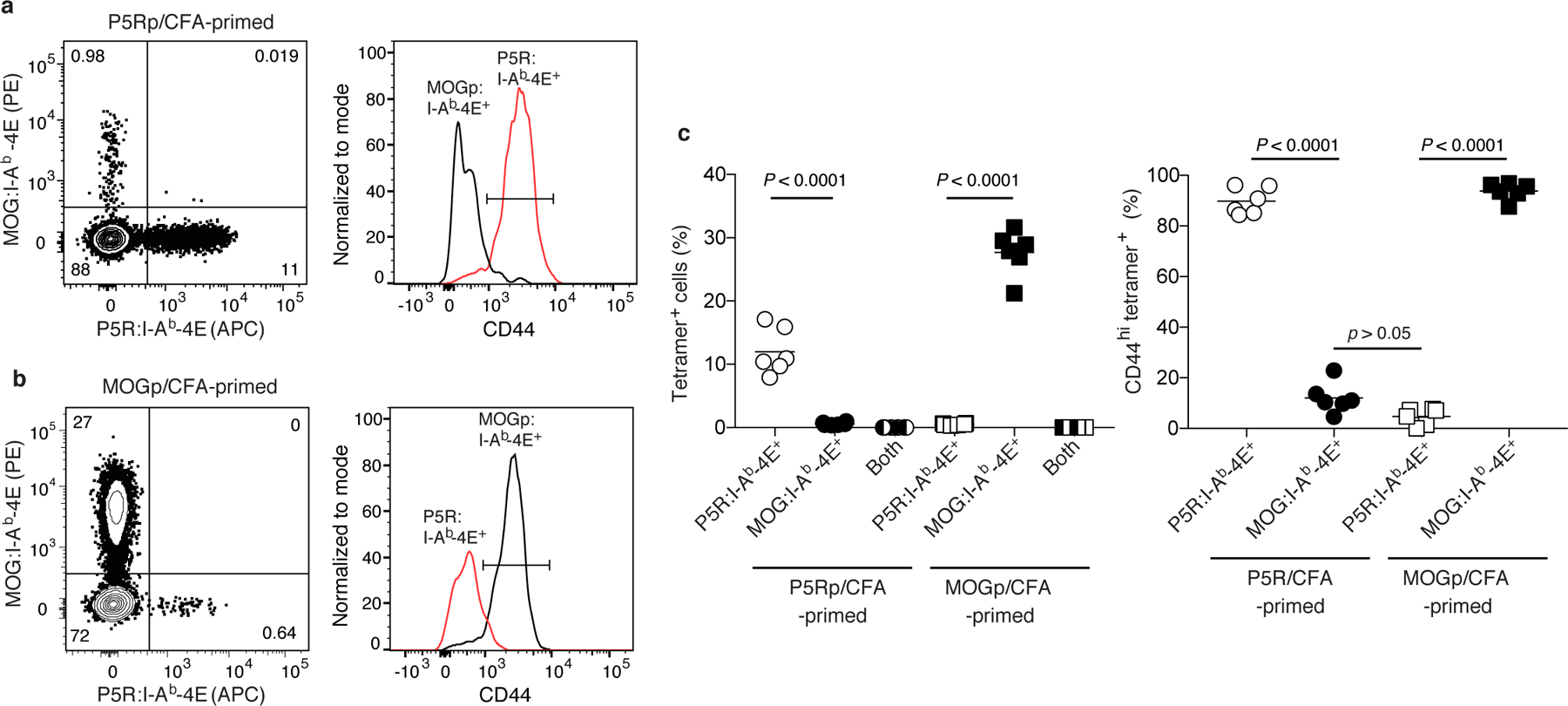
Fig3. 多克隆CD4+效应T细胞p:I-Ab -4E四聚体的检测具有肽特异性
讨论
基于CD4亲和力增强型四聚体的技术突破,有望实现对传统方法难以检测的表位特异性CD4⁺ T细胞的精准解析。这种技术革新具有双重重要意义:其一,此类"隐形"T细胞群体可能携带独特的免疫调节功能或表位扩散潜力,其功能特征显著区别于传统p:MHC II类四聚体可检出的T细胞库,为解析免疫应答的异质性提供了全新维度;其二,本研究构建的肽段特异性、CD4结合增强型MHC II类分子平台,为开发新一代免疫调节疗法奠定了分子工具基础。相较于天然MHC II类分子,这类工程化蛋白可通过调控CD4共刺激信号阈值,实现对抗原特异性T细胞的靶向激活或功能抑制,在自身免疫病治疗、肿瘤免疫治疗及疫苗效力优化等领域展现出转化医学潜力。这一技术体系不仅拓展了适应性免疫应答的研究边界,更可能催生基于T细胞表位精准调控的免疫干预新策略。
相关商品推荐
| 病原/肿瘤 | 产品名称 | 抗原 | 序列 | MHC | 位置 | 货号 |
| EBV | HLA-A*0201/YLELLVWRL-PE Labelled Tetramer | EBV.LMP1 | YLELLVWRL | HLA-A*0201 | 125-133 | UA089001 |
| EBV | HLA-A*0201/YLQQNWTL-PE Labelled Tetramer | EBV.LMP1 | YLQQNWTL | HLA-A*0201 | 159-167 | UA089003 |
| EBV | H-2Db(b)/RAHY-NIVTF-PE Labelled Tetramer | HPV16.E7 | RAHYNIVTF | H-2Db | 49-57 | UA089002 |
| HPV | H-2K(b)/EVYDFA-FRQL-PE Labelled Tetramer | HPV16.E6 | EVYDFARDL | H-2Kb | 48-57 | UA089004 |
| HPV | HLA-A*0201/KLP-DLCTL-PE Labelled Tetramer | HPV18.E6 | KLPDCTL | HLA-A*0201 | 13-21 | UA089005 |
| HPV | HLA-A*0201/KLTNT-GLYQL-PE Labelled Tetramer | HPV18.E6 | KLTNTGLYNL | HLA-A*0201 | 92-101 | UA089006 |
| HPV | HLA-A*0201/TLODIVIHL-PE Labelled Tetramer | HPV18.E7 | TLODIVIHL | HLA-A*0201 | 7~15 | UA089007 |
| HPV | HLA-A*0201/QFLNTL-FV-PE Labelled Tetramer | HPV18.E7 | QFLNTLFSV | HLA-A*0201 | 88-97 | UA089008 |
| HPV | HLA-A*1101/GVNHQLPAR-PE Labelled Tetramer | HPV18.E7 | GVNHQLPAR | HLA-A*1101 | 43-52 | UA089009 |
| Influenza A Virus | H-2D(b)/ASNENMETM-PE Labelled Tetramer | Flu.NP | ASNENMETM | H-2Db | 366-374 | UA089010 |
| Influenza A Virus | H-2K(d)/TYQR-TRALY-PE Labelled Tetramer | Flu.NP | TYQRTRALY | H-2Kd | 147-155 | UA089011 |
| Influenza A Virus | H-2D(b)/ASNEN-MDTM-PE Labelled Tetramer | Flu.NP | ASNENMDTM | H-2Db | 366-374 | UA089012 |
| LCMV | H-2D(b)/KAVYNFATM-PE Labelled Tetramer | GP 33 | KAVYNFATM | H-2Db | 33-41 | UA089013 |
| LCMV | H-2D(b)/FQPGQGFVK-PE Labelled Tetramer | LCMV NP | FQPGQGFVK | H-2Db | 396-404 | UA089014 |
| Tumor-related | HLA-A*1101/VVGADGVK-PE Labelled Tetramer | KRAS | VVGADGVK | HLA-A*1101 | 7~16 | UA089015 |
| Tumor-related | HLA-A*1101/VVGAGVGK-PE Labelled Tetramer | KRAS | VVGAGVGK | HLA-A*1101 | 7~16 | UA089016 |
| Tumor-related | HLA-A*0201/KLVVGAGV-PE Labelled Tetramer | KRAS | KLVVGAGV | HLA-A*0201 | 5~14 | UA089017 |
| Tumor-related | HLA-A*0201/SLLMWITQC-PE Labelled Tetramer | NY-ESO1 | SLLMWITQC | HLA-A*0201 | 157-165 | UA089018 |
| Melanoma | HLA-A*0201/LMWITQCFL-PE Labelled Tetramer | NY-ESO2 | LMWITQCFL | HLA-A*0201 | 159-167 | UA089019 |
| Melanoma | H-2Db(b)/MMFPNA-P1-PE Labelled Tetramer | WT1 | RMFPNAPL | H-2Db | 126-134 | UA089020 |
| Melanoma | HLA-A*0201/CMTWV-PE Labelled Tetramer | WT2 | CMTWVNMDM | HLA-A*0201 | 235-243 | UA089021 |
| Melanoma | HLA-A*1101/KTCQRKSF-PE Labelled Tetramer | WT3 | KTCQRKSF | HLA-A*1101 | 386-394 | UA089022 |
| Ovarian Cancer | H-2K(b)/SINFEKL-PE Labelled Tetramer | OVA | SINFEKL | H-2Kb | 257-264 | UA089023 |